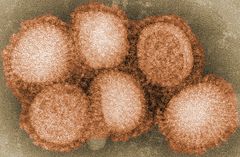
Virus de la grippe
Les virus de la grippe sont un ensemble de quatre espèces de virus à ARN monocaténaire de polarité négative distinguées chacune par un type antigénique particulier : le virus de la grippe A, le virus de la grippe B, le virus de la grippe C et le virus de la grippe D[1]. Parmi ces quatre types antigéniques, le type A est le plus dangereux, le type B présente moins de risques mais reste susceptible de provoquer des épidémies, et le type C n'est généralement associé qu'à des symptômes mineurs. Le type D est moins répandu que les autres et n'est pas connu pour provoquer des infections chez l'homme. Ces quatre virus ont un génome segmenté, à huit segments pour les deux premiers et à sept segments pour les deux derniers. La composition en acides aminés des virus de la grippe C et D est semblable à 50 %, taux semblable à celui observé entre les virus de la grippe A et B ; le taux de divergence entre les virus A et B d'une part et les virus C et D d'autre part est en revanche bien plus élevé[2].
| Domaine | Riboviria |
|---|---|
| Embranchement | Negarnaviricota |
| Sous-embr. | Polyploviricotina |
| Classe | Insthoviricetes |
| Ordre | Articulavirales |
| Famille | Orthomyxoviridae |
Les antigènes, protéine de matrice (M1) et nucléoprotéine (NP), sont utilisés pour déterminer si un virus de la grippe est de type A, B, C ou D. La protéine M1 est nécessaire pour l'assemblage du virus et la nucléoprotéine intervient dans la transcription et la réplication virale[3],[4].
Des glycoprotéines se trouvent également à la surface de la membrane cellulaire. Les virus de la grippe A et B ont deux glycoprotéines : l'hémagglutinine (HA) et la neuraminidase (NA) ; les virus de la grippe C et D n'ont qu'une glycoprotéine, la glycoprotéine de fusion hémagglutinine-estérase (HEF)[5]. Ces glycoprotéines permettent la fixation et la fusion entre l'enveloppe virale et la membrane cellulaire. La fusion de ces membranes permet aux protéines et au génome du virus d'être libérés dans la cellule hôte, ce qui provoque alors l'infection[6]. Les types C et D sont les seuls virus de la grippe à exprimer une estérase. Cette enzyme est semblable à la neuraminidase produite par les virus de la grippe A et B en ce que ces deux enzymes détruisent les récepteurs des cellules hôtes
Les glycoprotéines peuvent subir des mutations (glissement antigénique) ou un réassortiment dans lesquels une nouvelle hémagglutinine ou une nouvelle neuraminidase sont produites (cassure antigénique). Les virus de la grippe C et D ne peuvent donner lieu qu'à un glissement antigénique, alors que le virus de la grippe A connaît également des cassures antigéniques. Lorsque l'un ou l'autre de ces processus se produit, les anticorps produits par le système immunitaire de l'hôte ne protègent plus contre ces glycoprotéines modifiées. Pour cette raison, ces virus provoquent continuellement des infections.
Types, sous-types et souches
modifierLes quatre types de virus de la grippe correspondent chacun à une espèce de virus au sens de l'ICTV[7]. Chacune de ces espèces est à son tour subdivisée en sous-types, en sérotypes et en souches, dont la virulence et l'infectivité (en) varient sensiblement d'une espèce hôte à une autre.
| Types | Hôtes | Sous-types |
|---|---|---|
| Virus de la grippe A | Humains, porcs, oiseaux, chevaux, chauves-souris | H1N1, H1N2, H2N2, H3N1, H3N2, H3N8, H5N1, H5N2, H5N3, H5N8, H5N9, H7N1, H7N2, H7N3, H7N4, H7N7, H7N9, H9N2, H10N7 |
| Virus de la grippe B | Humains, phoques | Victoria, Yamagata |
| Virus de la grippe C | Humains, porcs, chiens | |
| Virus de la grippe D | Porcs, bovins |
Virus de la grippe A
modifierLes virus de la grippe A sont classés en fonction des glycoprotéines de leur enveloppe virale, l'hémagglutinine et la neuraminidase, ce qui identifiée des sous-types notés HxNy, où H identifie l'hémagglutinine et N identifie la neuraminidase, avec x variant de 1 à 18 et y variant de 1 à 11. Ce sont les plus virulents de virus de la grippe, les suivants ayant été observés chez les humains :
- le sous-type H1N1, à l'origine de l'épidémie de « grippe espagnole » de 1918, de « grippe russe » de 1977 et de « grippe porcine » de 2009[8] ;
- le sous-type H2N2, à l'origine de l'épidémie de « grippe asiatique » de 1956-1958 ;
- le sous-type H3N2, à l'origine de la « grippe de Hong Kong » de 1968-1969 ;
- le sous-type H5N1, à l'origine de la « grippe aviaire » de 2006, et considéré comme une menace de pandémie[9] ;
- le sous-type H7N7, qui présente un risque zoonotique particulièrement élevé[10] ;
- les sous-types H7N2, H7N3[11], H9N2 et H10N7[12] ont été sporadiquement observés chez des humains ;
- le sous-type H3N8 a pu être impliqué dans l'épidémie de 1889-1890[13], qui a tué plus d'un million de personnes.
Au sein de ces sous-types, on distingue également des souches et des variantes, identifiées selon une nomenclature indiquant respectivement le type, l'origine géographique, le numéro de la souche et l'année d'identification, par exemple :
- A/Fujian/411/2002 (H3N2) ;
- A/Brisbane/59/2007 (H1N1).
Les souches à l'origine de l'épidémie de « grippe aviaire » de 2006 sont généralement désignées par HPAI H5N1, signifiant Highly Pathogenic Avian Influenza[14],[15].
Virus de la grippe B
modifierLe virus de la grippe B est presque exclusivement humain, et moins répandu que le virus de la grippe A. Le seul hôte non humain connu pour héberger ce virus est le phoque[16]. Le virus de la grippe B mute deux à trois fois moins vite que celui de type A[17] et présente donc une variabilité génétique plus faible, avec un seul sérotype[18]. Pour cette raison, l'immunité contre les virus de la grippe B est généralement atteinte à un âge assez jeune. Le taux de mutation n'est cependant pas nul, et deux sous-types de virus de la grippe B sont clairement identifiés, appelés Victoria et Yamagata[19], de sorte que l'immunité ne peut être totale ni permanente. Néanmoins, l'absence d'hôtes non humains écarte la possibilité de l'émergence d'une pandémie à virus de grippe B[20]. Les variantes de ce virus sont identifiées par une nomenclature semblable à celle du virus de la grippe A, par exemple :
- B/Victoria/504/2000 ;
- B/Yamagata/16/88.
Virus de la grippe C
modifierLe virus de la grippe C infecte les humains et les porcs, et est susceptible de provoquer des affections sévères ainsi que des épidémies localisées[21]. Ce type de virus de la grippe est cependant moins courant que les autres et semble provoquer essentiellement des affections bénignes chez l'enfant[22],[23].
Virus de la grippe D
modifierLes premiers virus de la grippe D ont été isolés en 2011[24]. Ce type semble avoir divergé des virus de la grippe C il y a plusieurs centaines d'années[25]. On en connaît au moins deux souches[26]. Il semble infecter avant tout les bovins, mais a également été observé chez le porc.
Génome et structure
modifierLes virus de la grippe appartiennent à l'ordre des Articulavirales, de sorte qu'ils présentent un génome segmenté, c'est-à-dire divisé en molécules d'acides nucléiques distinctes, en l'occurrence de sept à huit segments d'ARN monocaténaire de polarité négative. La longueur totale de ce génome est comprise entre 12 000 et 15 000 nucléotides, avec, par exemple pour le virus de la grippe A (H1N1), une distribution du type :
| Segment d'ARN | Protéine(s) | Nucléotides | Acides aminés |
|---|---|---|---|
| PB1 | Polymérase basique 1 | 2300 à 2500 | 757 + 87 (F2) |
| PB2 | Polymérase basique 2 | 2300 à 2500 | 759 |
| PA | Polymérase acide | 2200 à 2300 | 716 |
| HA | Hémagglutinine | 1700 à 1800 | 550 |
| NP | Nucléoprotéine | 1500 à 1600 | 498 |
| NA | Neuraminidase | 1400 à 1500 | 454 |
| M | Protéines membranaires | 1000 à 1100 | 252 + 97 |
| NS | Protéines non structurelles | 800 à 900 | 230 + 121 |
Parmi les segments ci-dessus, les deux plus importants sont PB1 et HA. PB1 donne une ARN polymérase ARN-dépendante essentielle à la virulence du virus, tandis que HA donne l'antigène glycoprotéique de surface (l'hémagglutinine) déterminant sa transmissibilité.
La séquence du génome présente des séquences terminales répétées à chaque extrémité. Les séquences répétées à l'extrémité 5' ont une longueur de 12 ou 13 nucléotides, celles de l'extrémité 3' ont une longueur de 9 à 11 nucléotides. Le virus de la grippe A produit une protéine PB1-F2 à partir d'un cadre de lecture ouvert alternatif du segment PB1, tandis que les segments M et NS produisent deux protéines chacun, notées M1, M2, NS1 et NS2, par épissage alternatif[27].
-
Structure générique d'un virus de la grippe A indiquant l'agencement de 10 protéines virales. La protéine PB1-F2 et les huit segments d'ARN ne sont pas représentés sur ce schéma.
Les virions du virus de la grippe A ont un diamètre de 80 à 120 nm et forment généralement un ensemble de particules à la fois ellipsoïdales, bacilliformes et filamenteuses[28],[29],[30]. Leur génome a une longueur totale de 13,5 kilobases distribuées sur huit segments d'ARN monocaténaire de polarité négative qui codent 11 protéines, appelées HA, NA, NP, M1, M2, NS1, NEP, PA, PB1, PB1-F2 et PB2[31]. L'hémagglutinine HA et la neuraminidase NA sont les mieux caractérisées d'entre elles. Ce sont de grosses glycoprotéines situées sur la surface des virions. La neuraminidase est une enzyme intervenant dans le bourgeonnement des virions nouvellement formés dans les cellules infectées, tandis que l'hémagglutinine est une lectine intervenant dans la liaison du virus à la cellule hôte et dans l'injection du génome viral dans cette dernière[32]. L'hémagglutinine et la neuraminidase sont les cibles des antiviraux[33]. Ces protéines sont des antigènes reconnus par les anticorps de l'organisme[34].
Les virions sont pléomorphes, avec une enveloppe généralement ellipsoïde de 80 à 120 nm de diamètre et peuvent former des filaments atteignant 20 μm de long[29]. L'enveloppe est hérissée d'environ 500 protubérances faisant saillie d'environ 10 à 14 nm hors de la surface du virus. Les molécules d'hémagglutinine sont séparées les unes des autres par des grappes de neuraminidase, avec un ratio d'environ 4 à 5 molécules de neuraminidase par molécule d'hémagglutinine.
La membrane virale, chargée de cholestérol et hérissée de glycoprotéines, renferme la nucléocapside, qui contient des nucléoprotéines de différentes tailles arrangées selon une disposition mal connue. Les protéines ribonucléaires sont filamenteuses, avec un diamètre de 9 à 15 nm et une longueur de 50 à 130 nm. Elles présentent une symétrie hélicoïdale.
Cycle de réplication
modifierLes mammifères infectés transmettent la grippe généralement par voie aérienne, par la toux ou ses éternuements expulsant des aérosols contenant le virus, tandis que les oiseaux contaminés la transmettent par leurs excréments. La grippe peut également être transmise par la salive, les sécrétions nasales, les selles et le sang. Les infections surviennent par contact avec ces fluides corporels ou avec des surfaces contaminées. Hors de l'hôte, les virus de la grippe peuvent rester infectieux pendant environ une semaine à la température corporelle humaine (37 °C), pendant plus de 30 jours à 0 °C et indéfiniment à très basses températures — comme les lacs du nord-est de la Sibérie. Ils peuvent être facilement inactivés par les désinfectants et les détergents[35].
Les virus se lient à une cellule par le biais d'interactions entre leur glycoprotéine d'hémagglutinine et les oses des acides sialiques situés à la surface des cellules épithéliales des poumons et de la gorge[36] (étape 1 sur le schéma du cycle de réplication). La cellule absorbe le virus par endocytose. Dans l'endosome acide, une partie de l'hémagglutinine fusionne l'enveloppe virale avec la membrane de la vacuole, libérant dans le cytoplasme les molécules d'ARN viral (ARNv), les protéines accessoires et l'ARN polymérase ARN-dépendante[37] (étape 2). Ces protéines et l'ARN viral forment un complexe transporté dans le noyau de la cellule, où l'ARN polymérase ARN-dépendante peut transcrire l'ARN complémentaire (ARNc), de sens positif[38] (étapes 3a et 3b). L'ARNc peut être exporté dans le cytoplasme pour y être traduit (étape 4), mais peut également demeurer dans le noyau. Les protéines virales nouvellement synthétisées peuvent être sécrétées par l'appareil de Golgi vers la surface de la cellule (dans le cas de la neuraminidase et de l'hémagglutinine, correspondant à l'étape 5b du schéma), soit être transportées vers le noyau pour s'y lier à l'ARNv et former de nouvelles particules de génome viral (étape 5a). Les autres protéines virales ont plusieurs actions dans la cellule hôte, notamment la dégradation de l'ARN messager cellulaire et l'utilisation des nucléotides libérés pour la synthèse de l'ARN viral, ce qui a également pour effet d'inhiber la traduction des ARN messagers de la cellule hôte[39].
Les ARNv de polarité négative qui forment le génome de futurs virus, l'ARN polymérase ARN-dépendante et les autres protéines virales sont assemblés en un virion. Les molécules d'hémagglutinine et de neuraminidase se regroupent en un renflement dans la membrane cellulaire. L'ARNv et les protéines structurelles virales quittent le noyau et s'insèrent dans le renflement membranaire. Le virus mature bourgeonne hors la cellule à l'intérieur d'une sphère de membrane phospholipidique de la cellule hôte, emportant l'hémagglutinine et la neuraminidase avec cette enveloppe membranaire[40]. Les virus adhèrent à la cellule par l'intermédiaire de l'hémagglutinine, de sorte que les virus matures se détachent de la cellule hôte où ils se sont formés une fois que leur neuraminidase a clivé les résidus d'acide sialique de la cellule hôte[36]. Cette dernière meurt après la libération des virions du virus grippal.
Les virus de la grippe, comme tous les Orthomyxoviridae, se répliquent donc dans le noyau cellulaire, caractéristique qu'ils partagent seulement avec les rétrovirus. Cela vient du fait qu'ils ne disposent pas de la machinerie enzymatique nécessaire à la production de leurs propres ARN messagers. Ils utilisent des ARN cellulaires comme amorce pour commencer la synthèse de l'ARN messager viral par un mécanisme appelé capture de coiffe (en)[41]. Parvenue dans le noyau, la protéine PB2 de l'ARN polymérase se lie à la coiffe de l'extrémité 5' d'un ARN pré-messager cellulaire. La protéine PA clive cet ARN près de son extrémité 5' et utilise ce fragment coiffé comme amorce pour transcrire le reste du génome de l'ARN viral en ARN messager viral[42]. Cette façon de procéder est nécessaire pour que l'ARN messager viral ait une coiffe afin de pouvoir être reconnu par un ribosome et être traduit en protéine.
L'ARN polymérase ARN-dépendante virale étant dépourvue de fonction de correction d'erreur, elle commet une erreur d'insertion de nucléotide toutes les 10 000 insertions environ, ce qui est à peu près la longueur du génome des virus de la grippe. Chaque nouveau virion contient ainsi statistiquement une mutation dans son génome[43]. La segmentation du génome en molécules d'ARN distinctes permet de mélanger (réassortir) les gènes lorsqu'au moins deux variétés de virus de la grippe infectent la même cellule. Il s'ensuit la formation d'un virus pourvu de propriétés différentes, pouvant par exemple permettre l'infection de nouveaux hôtes ou de se soustraire à l'immunité développée par l'hôte contre le précédent génome ; dans ce dernier cas, on parle de cassure antigénique[34].
Viabilité du virus et désinfection
modifierLes virus de la grippe chez les mammifères ont tendance à être labiles, mais peuvent rester infectieux plusieurs heures dans le mucus[44]. Le virus de la grippe aviaire peut rester infectieux pendant 100 jours dans l'eau distillée à température ambiante, et pendant 200 jours à 17 °C. Il est inactivé plus rapidement dans le fumier mais peut demeurer infectieux pendant deux semaines dans la matière fécale. Les virus de la grippe aviaire peuvent rester infectieux indéfiniment lorsqu'ils sont congelés[44].
Les virus de la grippe sont sensibles à l'eau de javel, à l'éthanol à 70 %, aux aldéhydes, aux oxydants et aux ammoniums quaternaires. Ils sont inactivés par une température de 56 °C pendant au moins 60 minutes, ainsi que par un pH acide inférieur à 2[44].
Vaccins et prévention
modifierIl existe des vaccins et des médicaments pour la prophylaxie et le traitement des infections par un virus grippal. Les vaccins sont constitués de virions inactivés ou atténués de sous-type H1N1 ou H3N2 du virus de la grippe A humain, ainsi que des variantes du virus de la grippe B. Dans la mesure où l'antigénicité des virus sauvages évolue constamment, les vaccins sont reformulés chaque année à partir de nouvelles souches. Cependant, lorsque l'antigénicité des virus sauvages ne correspond pas à celle des souches utilisées pour produire ces vaccins, ces derniers ne protègent pas contre ces virus, et lorsque les antigénicités correspondent, il peut également apparaître des mutants échappant à la couverture antigénique des vaccins.
Parmi les médicaments disponibles pour le traitement de la grippe humaine, l'amantadine et la rimantadine inhibent le décollement des virions par des cellules infectées en interférant avec la protéine de matrice M2, tandis que l'oseltamivir (commercialisé sous la marque Tamiflu), le zanamivir et le peramivir inhibent la libération des virions en interférant avec la neuraminidase ; le peramivir tend à générer moins de mutants que le zanamivir[45]
Notes et références
modifier- (en) « Types of Influenza Viruses », CDC (consulté le ).
- (en) Shuo Su Jiangsu, Xinliang Fu, Gairu Li, Fiona Kerlin et Michael Veit, « Novel Influenza D virus: Epidemiology, pathology, evolution and biological characteristics », Virulence, vol. 8, no 8, , p. 1580-1591 (PMID 28812422, PMCID 5810478, DOI 10.1080/21505594.2017.1365216, lire en ligne)
- (en) Ayub Ali, Roy T. Avalos, Evgeni Ponimaskin et Debi P. Nayak, « Influenza Virus Assembly: Effect of Influenza Virus Glycoproteins on the Membrane Association of M1 Protein », Journal of Virology, vol. 74, no 18, , p. 8709-8719 (PMID 10954572, PMCID 116382, DOI 10.1128/JVI.74.18.8709-8719.2000, lire en ligne)
- (en) Agustín Portela et Paul Digard, « The influenza virus nucleoprotein: a multifunctional RNA-binding protein pivotal to virus replication », Journal of General Virology, vol. 83, no 4, , p. 723-734 (PMID 11907320, DOI 10.1099/0022-1317-83-4-723, lire en ligne)
- (en) Qinshan Gao, Edward W. A. Brydon et Peter Palese, « A Seven-Segmented Influenza A Virus Expressing the Influenza C Virus Glycoprotein HEF », Journal of Virology, vol. 82, no 13, , p. 6419-6426 (PMID 18448539, PMCID 2447078, DOI 10.1128/JVI.00514-08, lire en ligne)
- (en) W. Weissenhorn, A. Dessen, L. J. Calder, S. C. Harrison, J. J. Skehel et D. C. Wiley, « Structural basis for membrane fusion by enveloped viruses », Molecular Membrane Biology, vol. 16, no 1, , p. 3-9 (PMID 10332732, DOI 10.1080/096876899294706, lire en ligne)
- (en) « Virus Taxonomy: 2018b Release », ICTV, (consulté le ).
- (en) Taia T. Wang et Peter Palese, « Unraveling the Mystery of Swine Influenza Virus », Cell, vol. 137, no 6, , p. 983-985 (PMID 19524497, DOI 10.1016/j.cell.2009.05.032, lire en ligne)
- (en) J. K. Taubenberger et D. M. Morens, « Pandemic influenza – including a risk assessment of H5N1 », Revue Scientifique et Technique - Office International des Épizooties, vol. 28, no 1, , p. 187-202 (PMID 19618626, PMCID 2720801, lire en ligne)
- (en) Ron A. M. Fouchier, Peter M. Schneeberger, Frans W. Rozendaal, Jan M. Broekman, Stiena A. G. Kemink, Vincent Munster, Thijs Kuiken, Guus F. Rimmelzwaan, Martin Schutten, Gerard J. J. van Doornum, Guus Koch, Arnold Bosman, Marion Koopmans et Albert D. M. E. Osterhaus, « Avian influenza A virus (H7N7) associated with human conjunctivitis and a fatal case of acute respiratory distress syndrome », Proceedings of the National Academy of Sciences of the United States of America, vol. 101, no 5, , p. 1356-1361 (PMID 14745020, PMCID 337057, DOI 10.1073/pnas.0308352100, Bibcode 2004PNAS..101.1356F, lire en ligne)
- (en) « Protecting Poultry Workers from Avian Influenza (Bird Flu) », NIOSH, (DOI 10.26616/NIOSHPUB2008128, consulté le ).
- (en) P. F. Wright, G. Neumann et Y. Kawaoka, « 41-Orthomyxoviruses », revu par D. M. Knipe et P. M. Howley, Fields Virology, 1, 6e édition, Wolters Kluwer/Lippincott Williams & Wilkins, Philadelphie, 2013, p. 1201. (ISBN 978-1-4511-0563-6)
- (en) Alain-Jacques Valleron, Anne Cori, Sophie Valtat, Sofia Meurisse, Fabrice Carrat et Pierre-Yves Boëlle, « Transmissibility and geographic spread of the 1889 influenza pandemic », Proceedings of the National Academy of Sciences of the United States of America, vol. 107, no 19, , p. 8778-8781 (PMID 20421481, PMCID 2889325, DOI 10.1073/pnas.1000886107, JSTOR 25681493, Bibcode 2010PNAS..107.8778V, lire en ligne)
- (en) Siamak Zohari, Peter Gyarmati, Peter Thorén, György Czifra, Caroline Bröjer, Sándor Belá et kMikael Berg, « Genetic characterization of the NS gene indicates co-circulation of two sub-lineages of highly pathogenic avian influenza virus of H5N1 subtype in Northern Europe in 2006 », Virus Genes, vol. 36, no 1, , p. 117-125 (PMID 18172752, DOI 10.1007/s11262-007-0188-7, lire en ligne)
- (en) István Kiss, Péter Gyarmati, Siamak Zohari, Karin Wilbe Ramsay, Giorgi Metreveli, Elisabeth Weiss, Maria Brytting, Marielle Stivers, Sofia Lindström, Ake Lundkvist, Kirill Nemirov, Peter Thorén, Mikael Berg, György Czifra et Sándor Belák, « Molecular characterization of highly pathogenic H5N1 avian influenza viruses isolated in Sweden in 2006 », Virology Journal, vol. 5, , article no 113 (PMID 18837987, PMCID 2569034, DOI 10.1186/1743-422X-5-113, lire en ligne)
- (en) A. D. M. E. Osterhaus, G. F. Rimmelzwaan, B. E. E. Martina, T. M. Bestebroer et R. A. M. Fouchier, « Influenza B Virus in Seals », Science, vol. 288, no 5468, , p. 1051-1053 (PMID 10807575, DOI 10.1126/science.288.5468.1051, Bibcode 2000Sci...288.1051O, lire en ligne)
- (en) Eri Nobusawa et Katsuhiko Sato, « Comparison of the Mutation Rates of Human Influenza A and B Viruses », Journal of Virology, vol. 80, no 7, , p. 3675-3678 (PMID 16537638, PMCID 1440390, DOI 10.1128/JVI.80.7.3675-3678.2006, lire en ligne)
- (en) Alan J. Hay, Victoria Gregory, Alan R. Douglas et Yi Pu Lin, « The evolution of human influenza viruses », Philosophical Transactions of the Royal Society B, vol. 356, no 1416, , p. 1861-1870 (PMID 11779385, PMCID 1088562, DOI 10.1098/rstb.2001.0999, lire en ligne)
- (en) Barbara Biere, Bettina Bauer et Brunhilde Schweiger, « Differentiation of Influenza B Virus Lineages Yamagata and Victoria by Real-Time PCR », Journal of Microbial Microbiology, vol. 48, no 4, , p. 1425-1427 (PMID 20107085, PMCID 2849545, DOI 10.1128/JCM.02116-09, lire en ligne)
- (en) Maria C. Zambon, « Epidemiology and pathogenesis of influenza », Journal of Antimicrobial Chemotherapy, vol. 44, no 2, , p. 3-9 (PMID 10877456, DOI 10.1093/jac/44.suppl_2.3, lire en ligne)
- (en) Y. Matsuzaki, K. Sugawara, K. Mizuta, E. Tsuchiya, Y. Muraki, S. Hongo, H. Suzuki et K. Nakamura, « Antigenic and Genetic Characterization of Influenza C Viruses Which Caused Two Outbreaks in Yamagata City, Japan, in 1996 and 1998 », Journal of Clinical Microbiology, vol. 40, no 2, , p. 422-429 (PMID 11825952, PMCID 153379, DOI 10.1128/JCM.40.2.422-429.2002, lire en ligne)
- (en) Yoko Matsuzaki, Noriko Katsushima, Yukio Nagai, Makoto Shoji, Tsutomu Itagaki, Michiyo Sakamoto, Setsuko Kitaoka, Katsumi Mizuta et Hidekazu Nishimura, « Clinical Features of Influenza C Virus Infection in Children », The Journal of Infectious Diseases, vol. 193, no 9, , p. 1229-1235 (PMID 16586359, DOI 10.1086/502973, JSTOR 30086532, lire en ligne)
- (en) Susumu Katagiri, Akiko Ohizumi et Morio Homma, « An Outbreak of Type C Influenza in a Children's Home », The Journal of Infectious Diseases, vol. 148, no 1, , p. 51-56 (PMID 6309999, DOI 10.1093/infdis/148.1.51, JSTOR 30129337, lire en ligne)
- (en) Ben M. Hause, Mariette Ducatez, Emily A. Collin, Zhiguang Ran, Runxia Liu, Zizhang Sheng, Anibal Armien, Bryan Kaplan, Suvobrata Chakravarty, Adam D. Hoppe, Richard J. Webby, Randy R. Simonson et Feng Li, « Isolation of a Novel Swine Influenza Virus from Oklahoma in 2011 Which Is Distantly Related to Human Influenza C Viruses », PLoS Pathogens, vol. 9, no 2, , article no e1003176 (PMID 23408893, PMCID 3567177/, DOI 10.1371/journal.ppat.1003176, lire en ligne)
- (en) Zizhang Sheng, Zhiguang Ran, Dan Wang, Adam D. Hoppe, Randy Simonson, Suvobrata Chakravarty, Ben M. Hause et Feng Li, « Genomic and evolutionary characterization of a novel influenza-C-like virus from swine », Archives of Virology, vol. 159, no 2, , p. 249-255 (PMID 23942954, PMCID 5714291, DOI 10.1007/s00705-013-1815-3, lire en ligne)
- (en) Emily A. Collin, Zizhang Sheng, Yuekun Lang, Wenjun Ma, Ben M. Hause et Feng Li, « Cocirculation of Two Distinct Genetic and Antigenic Lineages of Proposed Influenza D Virus in Cattle », Journal of Virology, vol. 89, no 2, , p. 1036-1042 (PMID 25355894, PMCID 4300623, DOI 10.1128/JVI.02718-14, lire en ligne)
- (en) Nicole M. Bouvier et Peter Palese, « The biology of influenza viruses », Vaccine, vol. 24, no 4, , D49-D53 (PMID 19230160, PMCID 3074182, DOI 10.1016/j.vaccine.2008.07.039, lire en ligne)
- (en) Matthew D. Badham et Jeremy S. Rossman, « Filamentous Influenza Viruses », Current Clinical Microbiology Reports, vol. 3, no 3, , p. 155-161 (PMID 28042529, PMCID 5198887, DOI 10.1007/s40588-016-0041-7, lire en ligne)
- (en) Takeshi Noda, « Native morphology of influenza virions », Frontiers in Microbiology, vol. 2, , article no 269 (PMID 22291683, PMCID 3249889, DOI 10.3389/fmicb.2011.00269, lire en ligne)
- (en) Bernadeta Dadonaite, Swetha Vijayakrishnan, Ervin Fodor, David Bhella et Edward C. Hutchinson, « Filamentous influenza viruses », Journal of General Virology, vol. 97, no 8, , p. 1755-1764 (PMID 27365089, DOI 10.1099/jgv.0.000535, Bibcode 5935222, lire en ligne)
- (en) Elodie Ghedin, Naomi A. Sengamalay, Martin Shumway, Jennifer Zaborsky, Tamara Feldblyum, Vik Subbu, David J. Spiro, Jeff Sitz, Hean Koo, Pavel Bolotov, Dmitry Dernovoy, Tatiana Tatusova, Yiming Bao, Kirsten St George, Jill Taylor, David J. Lipman, Claire M. Fraser, Jeffery K. Taubenberger et Steven L. Salzberg, « Large-scale sequencing of human influenza reveals the dynamic nature of viral genome evolution », Nature, vol. 437, no 7062, , p. 1162-1166 (PMID 16208317, DOI 10.1038/nature04239, Bibcode 2005Natur.437.1162G, lire en ligne)
- (en) Yasuo Suzuki, « Sialobiology of Influenza: Molecular Mechanism of Host Range Variation of Influenza Viruses », Biological and Pharmaceutical Bulletin, vol. 28, no 3, , p. 399-408 (PMID 15744059, DOI 10.1248/bpb.28.399, lire en ligne)
- (en) J. C. Wilson et M. von Itzstein ., « Recent Strategies in the Search for New Anti-Influenza Therapies », Current Drug Targets, vol. 4, no 5, , p. 389-408 (PMID 12816348, DOI 10.2174/1389450033491019, lire en ligne)
- (en) Maurice R. Hilleman, « Realities and enigmas of human viral influenza: pathogenesis, epidemiology and control », Vaccine, vol. 20, nos 25-26, , p. 3068-3087 (PMID 12163258, DOI 10.1016/S0264-410X(02)00254-2, lire en ligne)
- (en) D. L. Suarez, E. Spackman, D. A. Senne, L. Bulaga, A. C. Welsch et K. Froberg, « The Effect of Various Disinfectants on Detection of Avian Influenza Virus by Real Time RT-PCR », Avian Diseases, vol. 47, no 3, , p. 1091-1095 (PMID 14575118, DOI 10.1637/0005-2086-47.s3.1091, lire en ligne)
- (en) Ralf Wagner, Mikhail Matrosovich et Hans‐Dieter Klenk, « Functional balance between haemagglutinin and neuraminidase in influenza virus infections », Reviews in Medical Virology, vol. 12, no 3, , p. 159-166 (PMID 11987141, DOI 10.1002/rmv.352, lire en ligne)
- (en) Melike Lakadamyali, Michael J. Rust, Hazen P. Babcock et Xiaowei Zhuang, « Visualizing infection of individual influenza viruses », Proceedings of the National Academy of Sciences of the United States of America, vol. 100, no 16, , p. 9280–9285 (PMID 12883000, PMCID 170909, DOI 10.1073/pnas.0832269100, Bibcode 2003PNAS..100.9280L, lire en ligne)
- (en) Jerome F. Cros et Peter Palese, « Trafficking of viral genomic RNA into and out of the nucleus: influenza, Thogoto and Borna disease viruses », Virus Research, vol. 95, nos 1-2, , p. 3-12 (PMID 12921991, DOI 10.1016/S0168-1702(03)00159-X, lire en ligne)
- (en) John C. Kash, Alan G. Goodman, Marcus J. Korth et Michael G. Katze, « Hijacking of the host-cell response and translational control during influenza virus infection », Virus Research, vol. 119, no 1, , p. 111-120 (PMID 16630668, DOI 10.1016/j.virusres.2005.10.013, lire en ligne)
- (en) Debi P.Nayak, Eric Ka-Wai Hui et Subrata Barman, « Assembly and budding of influenza virus », Virus Research, vol. 106, no 2, , p. 147-165 (PMID 15567494, DOI 10.1016/j.virusres.2004.08.012, lire en ligne)
- (en) « Cap snatching », sur ExPASy, Institut suisse de bioinformatique (consulté le ).
- (en) Alexandre Dias, Denis Bouvier, Thibaut Crépin, Andrew A. McCarthy, Darren J. Hart, Florence Baudin, Stephen Cusack et Rob W. H. Ruigrok, « The cap-snatching endonuclease of influenza virus polymerase resides in the PA subunit », Nature, vol. 458, no 7240, , p. 914-918 (PMID 19194459, DOI 10.1038/nature07745, Bibcode 2009Natur.458..914D, lire en ligne)
- (en) J W Drake, « Rates of spontaneous mutation among RNA viruses. », Proceedings of the National Academy of Sciences of the United States of America, vol. 90, no 9, , p. 4171-4175 (PMID 8387212, PMCID 46468, DOI 10.1073/pnas.90.9.4171, Bibcode 1993PNAS...90.4171D, lire en ligne)
- (en) Anna Rovid Spickler, « Influenza » [PDF], sur The Center in Food Security & Public Health, Université d'État de l'Iowa, (consulté le ).
- (en) Yoshiyuki Suzuki, « Natural Selection on the Influenza Virus Genome », Molecular Biology and Evolution, vol. 23, no 10, , p. 1902-1911 (PMID 16818477, DOI 10.1093/molbev/msl050, lire en ligne)